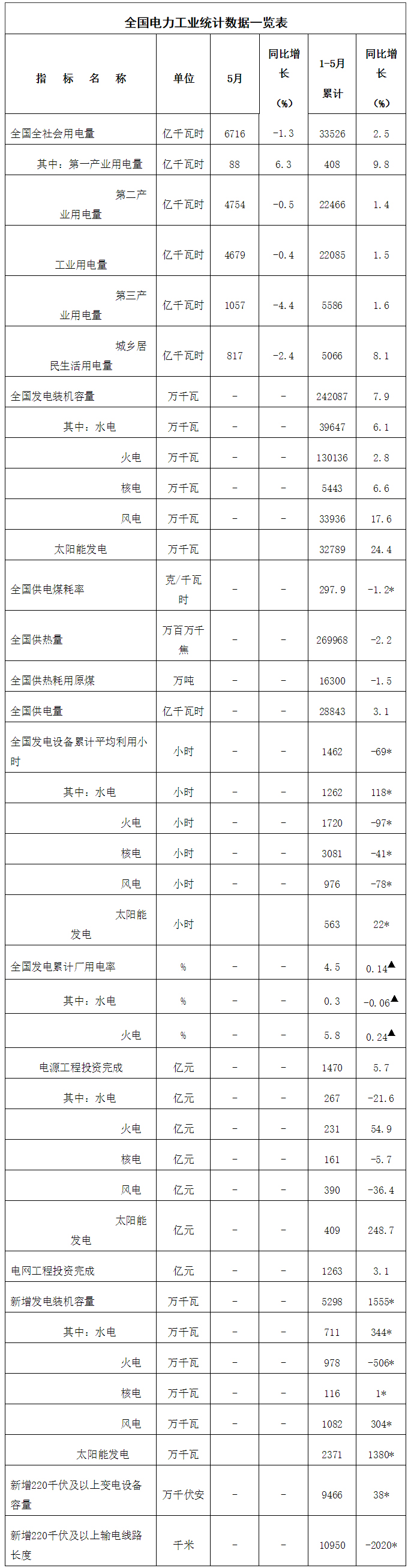
来源:生物谷原创 | 2022-06-21 05:40:45 |
单细胞RNA测序研究表明,机体总mRNA含量或与肿瘤表型直接相关,然而,截止到目前为止,技术和分析方面的挑战严重阻碍了科学家们对总mRNA含量进行大规模的泛癌分析。
近日,一篇发表在国际杂志Nature Biotechnology上题为“Estimation of tumor cell total mRNA expression in 15 cancer types predicts disease progression”的研究报告中,来自德克萨斯大学等机构的科学家们通过研究开发了一种新方法,其能对来自患者机体肿瘤样本中的肿瘤特异性总mRNA水平进行定量分析,这些肿瘤样本中包含癌症和非癌症细胞,利用这种新技术对来自15种癌症类型超过6500份患者的肿瘤样本进行分析,研究人员发现,癌细胞中高水平的mRNA或与患者生存率下降直接相关。
本文研究结果表明,这种新型的计算方法或能对来自肿瘤样本中的肿瘤特异性总mRNA水平进行大规模分析,从而作为多种类型癌症的潜在预后生物标志物。研究者Wenyi Wang说道,单细胞测序研究结果表明,癌细胞中总mRNA含量与肿瘤的生物学特征直接相关,但使用单细胞方法来分析大规模患者群体或许是不可行的。基于这项最新研究,研究人员提出了一种新型的数学去卷积技术,即利用广泛可用的大量肿瘤测序数据来大规模研究癌症的重要生物学特性。
 (资料图)
(资料图)
单细胞测序手段能对样本中数千个单个细胞进行分析,而批量测序则能在更多细胞中产生一张肿瘤的整体图谱,由于肿瘤样本中包含了多种癌细胞和非癌细胞的混合体,因此研究人员还需要额外的步骤来从批量测序数据中分离出癌症特定的信息。去卷积是一种能将批量测序数据分离成不同的组成部分的计算技术,这项研究中,研究人员首先报道了一种新型的去卷积方法,其能定量来自大量测序数据中的总的肿瘤特异性mRNA水平,从而为单细胞分析提供一种可扩展的补充。
为了开发这种去卷积工具,研究人员对来自患有4种不同癌症类型的10名患者机体48,913细胞的单细胞测序数据进行分析,随后将这些数据汇总在一起,就好像在大量样本中一样,从而就能帮助识别出癌细胞和非癌细胞中总mRNA水平的差异,同时还能促使他们进一步分析大量肿瘤样本中的这些差异。当利用癌细胞系证实了这种方法后,研究人员利用来自四个大型队列的6580名患者的大量测序数据量化了肿瘤特异性的mRNA水平,由于大批量测序已经被常规使用了很多年,因此研究人员还能将总mRNA水平与这些患者的长期临床数据进行比较。
在一项泛癌分析中,研究人员发现,高水平的总肿瘤特异性mRNA水平或与患者无进展生存期及总体生存期减少相关。有趣的是,这种关联性依赖于癌症发生的阶段,在某些队列中,观察癌症的特定阶段或许就能阐明高水平的mRNA反而与患者预后改善直接相关,由于早期和晚期癌症有着不同的治疗方案,因此研究人员建议,总mRNA水平或能被潜在用来预测患者的预后以及对某些疗法的反应。
图片来源:https://pubmed.ncbi.nlm.nih.gov/35697807/
具体分析两个独立的乳腺癌患者队列,研究人员证实,较高的总mRNA水平或与化疗治疗的早期阶段患者预后的改善直接相关,相反,在这一队列中,携带较低总mRNA水平的早期阶段患者似乎从化疗中获益较少。这些研究发现必须通过更大规模的前瞻性试验来证实,但研究人员建议,肿瘤特异性的总mRNA水平或许能被调整为预后生物标志物来对高风险患者进行分层并指导患者的治疗选择。
从目前可用的临床工具来讲,研究人员知道,分析特定途径或一组基因的表达变化或许有望帮助指导患者的治疗,同时相关研究结果也强调,将转录组作为一个整体来看或许可能会更加强大。综上,本文研究结果表明,测定肿瘤细胞中细胞类型特异性的总mRNA表达或许能帮助预测肿瘤的表型以及临床治疗结果。(生物谷Bioon.com)
原始出处:
Cao, S., Wang, J.R., Ji, S.et al.Estimation of tumor cell total mRNA expression in 15 cancer types predicts disease progression.Nat Biotechnol(2022). doi:10.1038/s41587-022-01342-x

2022-06-20 14:48:38

2022-06-20 14:36:36

2022-06-20 14:25:19

2022-06-20 14:02:57

2022-06-20 13:54:16
2022-06-17 16:02:08

2022-06-17 14:05:06

2022-06-17 11:28:44

2022-06-17 10:01:54

2022-06-17 09:59:32

2022-06-17 09:55:50

2022-06-17 09:51:44

2022-06-17 09:49:42

2022-06-17 09:48:49

2022-06-17 09:48:25

2022-06-17 09:31:54

2022-06-17 09:31:42

2022-06-17 09:29:34

2022-06-17 09:27:21

2022-06-17 09:24:45

2022-06-17 09:22:31

2022-06-17 09:19:46

2022-06-17 09:18:01

2022-06-17 09:13:58

2022-06-17 09:09:58
2022-06-17 09:06:31

2022-06-17 08:53:57

2022-06-17 08:50:50

2022-06-17 05:49:49

2022-06-17 05:49:38

2022-06-17 05:47:10

2022-06-17 05:46:07

2022-06-17 05:46:00

2022-06-17 05:45:50

2022-06-17 05:44:19

2022-06-17 05:43:09

2022-06-17 05:41:04

2022-06-17 05:37:12

2022-06-17 05:32:41

2022-06-17 05:32:21

2022-06-17 05:32:01

2022-06-17 05:31:37

2022-06-17 05:29:15

2022-06-16 22:59:17

2022-06-16 17:57:51

2022-06-16 17:53:11

2022-06-16 17:48:54

2022-06-16 17:39:38

2022-06-16 17:33:37

2022-06-16 17:30:22

2022-06-16 17:30:20

2022-06-16 17:27:31

2022-06-16 17:23:43

2022-06-16 17:20:58

2022-06-16 17:19:12

2022-06-16 17:16:51

2022-06-16 17:15:18

2022-06-16 17:12:34

2022-06-16 17:08:31

2022-06-16 17:05:44

2022-06-16 16:54:33

2022-06-16 16:49:31

2022-06-16 16:46:40

2022-06-16 16:41:47

2022-06-16 16:38:39

2022-06-16 16:36:07

2022-06-16 16:31:23

2022-06-16 16:29:20

2022-06-16 16:25:42

2022-06-16 16:23:25

2022-06-16 16:19:17

2022-06-16 16:13:48

2022-06-16 16:08:40

2022-06-16 16:02:42

2022-06-16 16:00:10

2022-06-16 15:37:06

2022-06-16 14:30:34

2022-06-16 13:13:43

2022-06-16 09:25:28

2022-06-16 09:20:59

2022-06-16 09:18:19

2022-06-16 09:12:53

2022-06-16 09:09:52

2022-06-16 09:06:38

2022-06-16 09:01:17

2022-06-16 08:59:43

2022-06-16 08:53:02

2022-06-16 08:49:39

2022-06-16 08:46:36

2022-06-16 08:43:30

2022-06-16 08:40:16

2022-06-16 06:44:28

2022-06-16 05:50:43

2022-06-16 05:49:55

2022-06-16 05:42:03

2022-06-16 05:40:51

2022-06-16 05:39:52

2022-06-16 05:39:44

2022-06-16 05:39:09

2022-06-16 05:36:24

2022-06-16 05:35:16

2022-06-16 05:34:45

2022-06-16 05:33:25

2022-06-16 05:31:22

2022-06-16 05:29:20
